plotconfusion
绘制分类混淆矩阵
语法
说明
plotconfusion( 绘制真实标签 targets,outputs)targets 和预测标签 outputs 的混淆矩阵。将标签指定为分类向量,或以一对 N (one-hot) 形式指定。
提示
不推荐将 plotconfusion 用于分类标签。请改用 confusionchart。
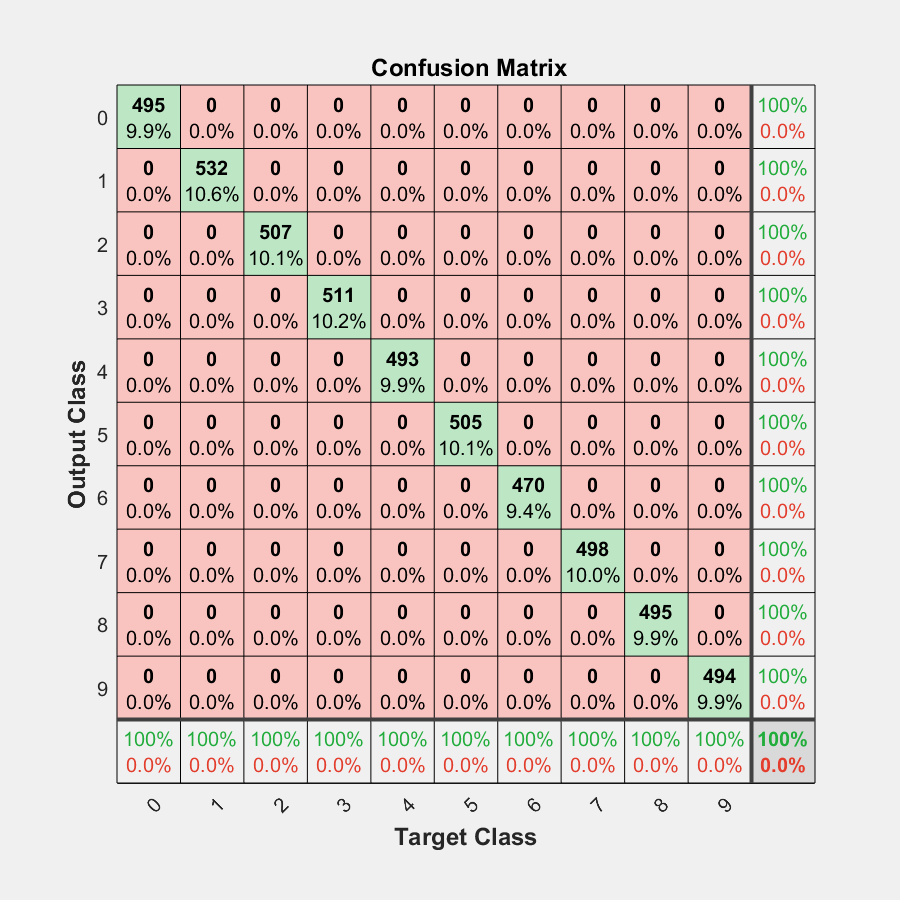
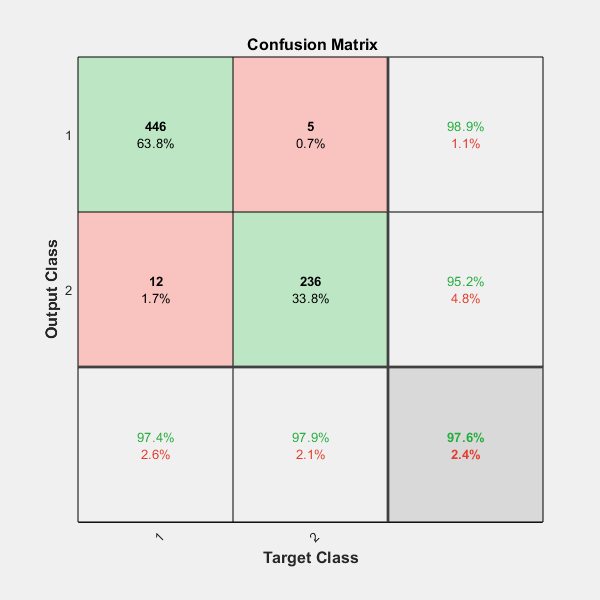
在混淆矩阵图上,行对应于预测类(输出类),列对应于真实类(目标类)。对角线上的单元格对应于正确分类的观测值。非对角线上的单元格对应于未正确分类的观测值。每个单元格中都显示观测值数目和观测值数目占总观测值数目的百分比。
绘图最右侧的列显示预测属于正确分类和未正确分类的每个类的示例占所有示例的百分比。这些度量通常分别称为精确度(或正预测值)和假发现率。绘图底部的行显示属于正确分类和未正确分类的每个类的示例占所有示例的百分比。这些度量通常分别称为召回率(或真正率)和假负率。绘图右下角的单元格显示整体准确度。
plotconfusion(targets1,outputs1,name1,targets2,outputs2,name2,...,targetsn,outputsn,namen) 在一个图窗中绘制多个混淆矩阵,并将 name 参量添加到对应绘图标题的开头。
示例
输入参数
版本历史记录
在 R2008a 中推出