使用深度学习进行波形分割
此示例说明如何使用递归深度学习网络和时频分析来分割人体心电图 (ECG) 信号。
简介
人类心脏的电活动可以作为相对于基线信号的振幅序列来测量。对于单个正常心跳周期,ECG 信号可分为以下几种心跳形态 [1]:
P 波 - 表示心房去极化的 QRS 复合波前的小偏转
QRS 复合波 - 心跳的最大振幅部分
T 波 - 表示心室复极化的 QRS 复合波后的小偏转
ECG 波形的这些区域的分割可作为基础测量数据用于评估人类心脏整体健康和异常状况 [2]。手动对 ECG 信号的每个区域进行注解可能是一项乏味且耗时的任务。信号处理和深度学习方法可能有助于简化注解并自动对感兴趣的区域进行注解。
此示例使用来自公开可用的 QT 数据库的 ECG 信号 [3] [4]。这些数据包括大约 15 分钟的 ECG 记录,采样率为 250 Hz,来自总共 105 名患者。为了获得每个记录,检查人员将两个电极放置在患者胸部的不同位置,以产生双通道信号。该数据库提供由自动专家系统生成的信号区域标签 [2]。此示例旨在使用深度学习解决方案根据采样所在的区域为每个 ECG 信号采样提供标签。这种为信号中的感兴趣区域加标签的过程通常称为波形分割。
为了训练深度神经网络来对信号区域进行分类,您可以使用长短期记忆 (LSTM) 网络。此示例说明如何使用信号预处理方法和时频分析来提高 LSTM 分割性能。具体而言,此示例使用傅里叶同步压缩变换来表示 ECG 信号的非平稳行为。
下载并准备数据
105 个双通道 ECG 信号的每个通道由自动专家系统独立标注并独立处理,总共 210 个 ECG 信号,它们与区域标签一起存储在 210 个 MAT 文件中。这些文件可在以下位置获得:https://www.mathworks.com/supportfiles/SPT/data/QTDatabaseECGData.zip。
将数据文件下载到您的临时目录中,临时目录的位置由 MATLAB® 的 tempdir 命令指定。如果您要将数据文件放在不同于 tempdir 的文件夹中,请在后续指令中更改目录名称。
% Download the data dataURL = 'https://www.mathworks.com/supportfiles/SPT/data/QTDatabaseECGData1.zip'; datasetFolder = fullfile(tempdir,'QTDataset'); zipFile = fullfile(tempdir,'QTDatabaseECGData.zip'); if ~exist(datasetFolder,'dir') websave(zipFile,dataURL); unzip(zipFile,tempdir); end
unzip 操作会在您的临时目录中创建 QTDatabaseECGData 文件夹,其中包含 210 个 MAT 文件。每个文件在变量 ecgSignal 中包含一个 ECG 信号,在变量 signalRegionLabels 中包含一个区域标签表。每个文件还在变量 Fs 中包含信号的采样率。在此示例中,所有信号的采样率均为 250 Hz。
创建一个信号数据存储来访问文件中的数据。此示例假设数据集已存储在临时目录中的 QTDatabaseECGData 文件夹下。如果不是这样,请更改下面代码中数据的路径。使用 SignalVariableNames 参数指定要从每个文件中读取的信号变量名称。
sds = signalDatastore(datasetFolder,'SignalVariableNames',["ecgSignal","signalRegionLabels"])
sds =
signalDatastore with properties:
Files:{
'/tmp/QTDataset/ecg1.mat';
'/tmp/QTDataset/ecg10.mat';
'/tmp/QTDataset/ecg100.mat'
... and 207 more
}
Folders: {'/tmp/QTDataset'}
AlternateFileSystemRoots: [0×0 string]
ReadSize: 1
SignalVariableNames: ["ecgSignal" "signalRegionLabels"]
ReadOutputOrientation: "column"
OutputDataType: "same"
OutputEnvironment: "cpu"
每次调用 read 函数时,数据存储都会返回一个包含 ECG 信号和区域标签表的二元素元胞数组。使用数据存储的 preview 函数,可以看到第一个文件的内容是长度为 225,000 个采样的 ECG 信号和一个包含 3385 个区域标签的表。
data = preview(sds)
data=2×1 cell array
{225000×1 double}
{ 3385×2 table }
查看区域标签表的前几行,注意观察是否每行都包含区域范围索引和区域类值(P、T 或 QRS)。
head(data{2}) ROILimits Value
__________ _____
83 117 P
130 153 QRS
201 246 T
285 319 P
332 357 QRS
412 457 T
477 507 P
524 547 QRS
使用 signalMask 对象可视化前 1000 个采样的标签。
M = signalMask(data{2});
plotsigroi(M,data{1}(1:1000))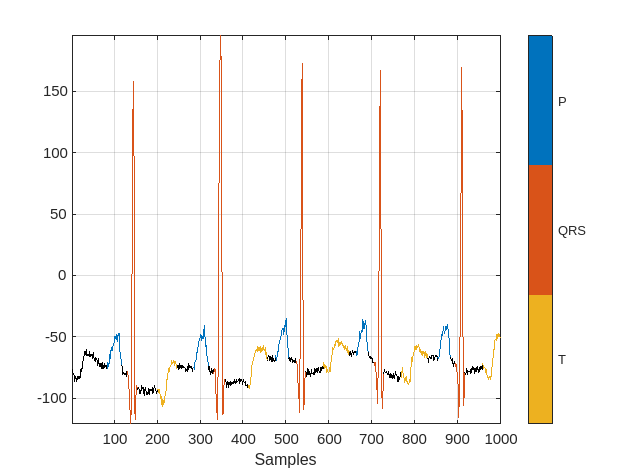
通常的机器学习分类过程如下:
将数据库分成训练数据集和测试数据集。
使用训练数据集训练网络。
使用经过训练的网络对测试数据集进行预测。
用 70% 的数据对网络进行训练,用剩余的 30% 对网络进行测试。
为了获得可重现的结果,请重置随机数生成器。使用 dividerand 函数获得随机索引来对文件进行乱序处理,使用 signalDatastore 的 subset 函数将数据分成训练数据存储和测试数据存储。
rng default
[trainIdx,~,testIdx] = dividerand(numel(sds.Files),0.7,0,0.3);
trainDs = subset(sds,trainIdx);
testDs = subset(sds,testIdx);在此分割问题中,LSTM 网络的输入是 ECG 信号,输出是与输入信号长度相同的标签序列或标签掩膜。网络任务是用信号采样所属区域的名称来标注每个信号采样。因此,有必要将数据集中的区域标签变换为序列,序列中的每个标签对应一个信号采样。使用变换后的数据存储和 getmask 辅助函数来变换区域标签。getmask 函数会添加一个标签类别 "n/a",用于标注不属于任何感兴趣区域的采样。
type getmask.mfunction outputCell = getmask(inputCell)
%GETMASK Convert region labels to a mask of labels of size equal to the
%size of the input ECG signal.
%
% inputCell is a two-element cell array containing an ECG signal vector
% and a table of region labels.
%
% outputCell is a two-element cell array containing the ECG signal vector
% and a categorical label vector mask of the same length as the signal.
% Copyright 2020 The MathWorks, Inc.
sig = inputCell{1};
roiTable = inputCell{2};
L = length(sig);
M = signalMask(roiTable);
% Get categorical mask and give priority to QRS regions when there is overlap
mask = catmask(M,L,'OverlapAction','prioritizeByList','PriorityList',[2 1 3]);
% Set missing values to "n/a"
mask(ismissing(mask)) = "n/a";
outputCell = {sig,mask};
end
预览变换后的数据存储,观察它是否返回长度相等的信号向量和标签向量。绘制分类封装向量的前 1000 个元素。
trainDs = transform(trainDs, @getmask); testDs = transform(testDs, @getmask); transformedData = preview(trainDs)
transformedData=1×2 cell array
{224993×1 double} {224993×1 categorical}
plot(transformedData{2}(1:1000))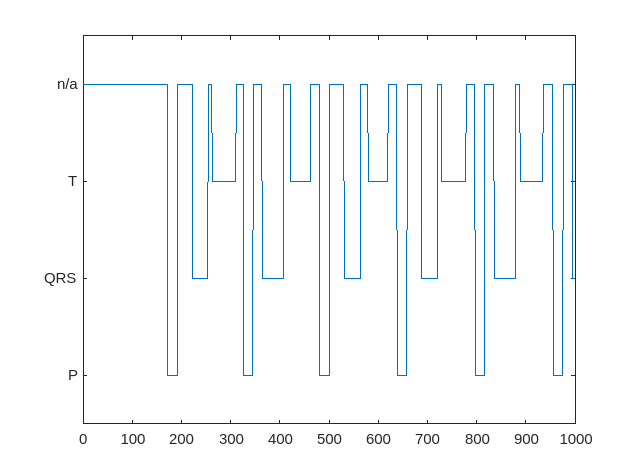
将非常长的输入信号传递给 LSTM 网络可能会导致估计性能下降和内存使用量过多。为了避免这些影响,请使用变换后的数据存储和 resizeData 辅助函数来拆分 ECG 信号及其对应的标签掩膜。该辅助函数会创建尽可能多的包含 5000 个采样的信号段,并丢弃其余采样。变换后的数据存储的输出预览显示,第一个 ECG 信号及其标签掩膜被分成了若干包含 5000 个采样的信号段。请注意,变换后的数据存储的预览仅显示 8 个元素,它们是在我们调用数据存储 read 函数时会生成的包含 floor(224993/5000) = 44 个元素的元胞数组的前 8 个元素。
trainDs = transform(trainDs,@resizeData); testDs = transform(testDs,@resizeData); preview(trainDs)
ans=8×2 cell array
{5000×1 double} {5000×1 categorical}
{5000×1 double} {5000×1 categorical}
{5000×1 double} {5000×1 categorical}
{5000×1 double} {5000×1 categorical}
{5000×1 double} {5000×1 categorical}
{5000×1 double} {5000×1 categorical}
{5000×1 double} {5000×1 categorical}
{5000×1 double} {5000×1 categorical}
选择训练网络或下载预训练网络
此示例的以下内容部分比较了三种不同的 LSTM 网络训练方法。由于数据集很大,每个网络的训练过程可能需要几分钟。如果您的机器同时有 GPU 和 Parallel Computing Toolbox™,则 MATLAB 会自动使用 GPU 以加快训练速度。否则将使用 CPU。
您可以跳过训练步骤,使用以下选择器下载预训练网络。如果您要在示例运行时训练网络,请选择 'Train Networks'。如果您要跳过训练步骤,请选择 'Download Networks',然后会有一个包含所有三个预训练网络 - rawNet、filteredNet 和 fsstNet- 的文件下载到您的临时目录中,其位置由 MATLAB® 的 tempdir 命令指定。如果要将下载的文件放在不同于 tempdir 的文件夹中,请在后续指令中更改目录名称。
actionFlag ="Train networks"; if actionFlag == "Download networks" Download the pre-trained networks dataURL = 'https://ssd.mathworks.com/supportfiles/SPT/data/QTDatabaseECGSegmentationNetworks.zip'; %#ok<*UNRCH> modelsFolder = fullfile(tempdir,'QTDatabaseECGSegmentationNetworks'); modelsFile = fullfile(modelsFolder,'trainedNetworks.mat'); zipFile = fullfile(tempdir,'QTDatabaseECGSegmentationNetworks.zip'); if ~exist(modelsFolder,'dir') websave(zipFile,dataURL); unzip(zipFile,fullfile(tempdir,'QTDatabaseECGSegmentationNetworks')); end load(modelsFile) rawNet = dag2dlnetwork(rawNet); filteredNet = dag2dlnetwork(filteredNet); fsstNet = dag2dlnetwork(fsstNet); end
下载的网络和新训练的网络之间的结果可能略有不同,因为网络是使用随机初始权重训练的。
将原始 ECG 信号直接输入 LSTM 网络
首先,使用来自训练数据集的原始 ECG 信号训练 LSTM 网络。
在训练前定义网络架构。指定大小为 1 的 sequenceInputLayer,以接受一维时间序列。使用 'sequence' 输出模式指定一个 LSTM 层,以便为信号中的每个采样提供分类。使用 200 个隐藏节点以获得最佳性能。指定输出大小为 4 的 fullyConnectedLayer,对每个波形类指定一个层。
layers = [ ... sequenceInputLayer(1) lstmLayer(200,'OutputMode','sequence') fullyConnectedLayer(4) softmaxLayer];
为训练过程选择选项,以确保获得良好的网络性能。有关每个参数的描述,请参阅 trainingOptions 文档。
options = trainingOptions('adam', ... 'MaxEpochs',10, ... 'MiniBatchSize',50, ... 'InitialLearnRate',0.01, ... 'LearnRateDropPeriod',3, ... 'LearnRateSchedule','piecewise', ... 'GradientThreshold',1, ... 'Plots','training-progress',... 'shuffle','every-epoch',... 'Verbose',0, ... 'Metrics','accuracy');
由于整个训练数据集可放入内存,因此,如果你有可用的 Parallel Computing Toolbox™,则可以使用数据存储的 tall 函数以并行方式变换数据,然后将其收集到工作区中。神经网络训练是迭代进行的。在每次迭代中,数据存储从文件中读取数据,变换数据,然后更新网络系数。如果数据可放入计算机的内存中,则将数据导入工作区可以加快训练速度,因为数据只需读取和变换一次。请注意,如果数据无法放入内存,您必须将数据存储传递给训练函数,并且在每轮训练中执行变换。
为训练集和测试集创建 tall 数组。根据您的系统,MATLAB 创建的并行池中的工作单元数量可能会有所不同。
tallTrainSet = tall(trainDs);
Starting parallel pool (parpool) using the 'Processes' profile ... Connected to parallel pool with 16 workers.
tallTestSet = tall(testDs);
现在调用 tall 数组的 gather 函数来计算整个数据集上的变换,并获得具有训练和测试信号及标签的元胞数组。
trainData = gather(tallTrainSet);
Evaluating tall expression using the Parallel Pool 'Processes': - Pass 1 of 1: Completed in 7.8 sec Evaluation completed in 7.9 sec
trainData(1,:)
ans=1×2 cell array
{5000×1 double} {5000×1 categorical}
testData = gather(tallTestSet);
Evaluating tall expression using the Parallel Pool 'Processes': - Pass 1 of 1: Completed in 1.6 sec Evaluation completed in 1.7 sec
训练网络
使用 trainnet 命令训练 LSTM 网络。
if actionFlag == "Train networks" rawNet = trainnet(trainData(:,1),trainData(:,2),layers,"crossentropy",options); end
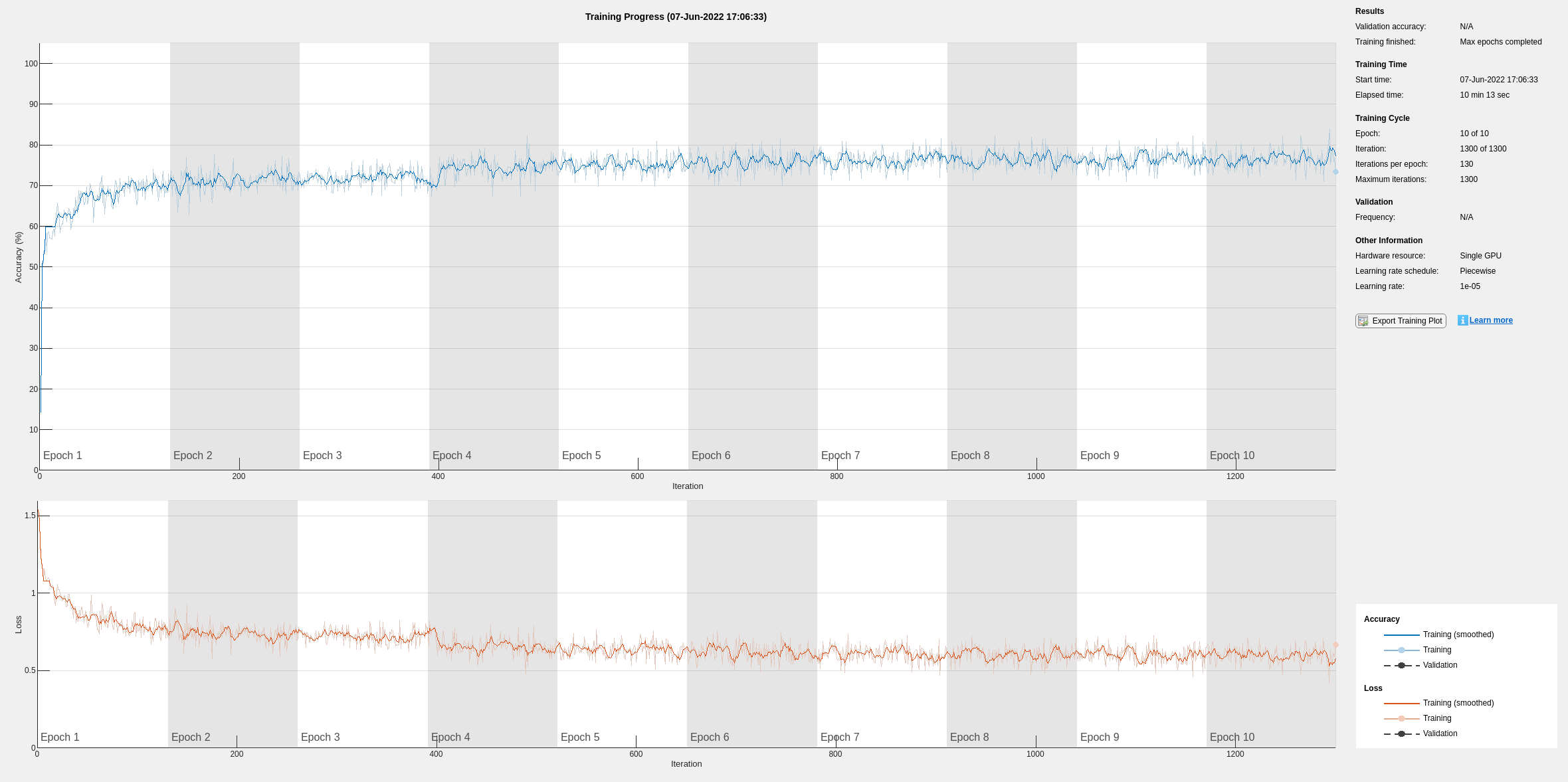
图窗中的训练准确度和损失子图会跟踪所有迭代的训练进度。使用原始信号数据,网络将大约 77% 的采样正确分类为 P 波、QRS 复合波、T 波或未标注区域 "n/a"。
对测试数据进行分类
使用经过训练的 LSTM 网络对测试数据进行分类。要使用多个观测值进行预测,请使用 minibatchpredict 函数。要将预测分数转换为标签,请使用 scores2label 函数。请指定小批量大小 50 以匹配训练选项。
classNames = categories(trainData{1,2});
scores = minibatchpredict(rawNet,testData(:,1),'MiniBatchSize',50,'UniformOutput',false);
predTest = scores2label(scores,classNames);混淆矩阵提供了一种直观的方式来可视化分类性能。使用 confusionchart 命令计算用于测试数据预测的总体分类准确度。对于每个输入,请将分类标签元胞数组转换为向量。指定行归一化显示,以每个类的采样百分比形式查看结果。
confusionchart(vertcat(testData{:,2}),vertcat(predTest{:}),'Normalization','row-normalized');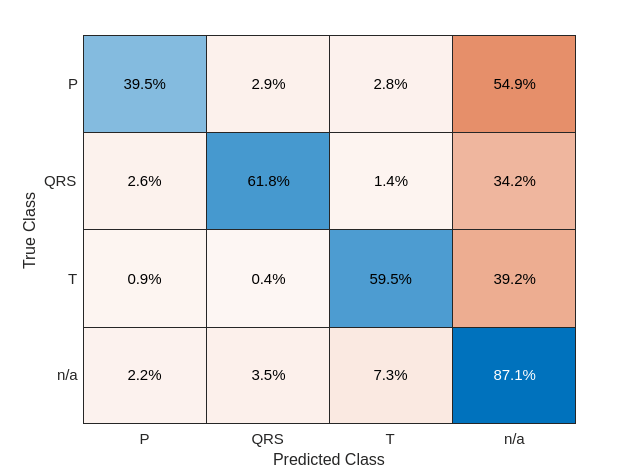
如果使用原始 ECG 信号作为网络的输入,则只有大约 60% 的 T 波采样、40% 的 P 波采样和 60% 的 QRS 复合波采样是正确的。为了提高性能,请在输入到深度学习网络之前应用一些 ECG 信号特征的知识,例如由患者呼吸运动引起的基线漂移。
应用滤波方法以消除基线漂移和高频噪声
这三种心跳形态占据不同频带。QRS 复合波的频谱通常以大约 10-25 Hz 为中心频率,并且其分量低于 40 Hz。发生 P 波和 T 波的频率甚至更低:P 波分量低于 20 Hz,T 波分量低于 10 Hz [5]。
基线漂移是由患者呼吸运动引起的低频 (< 0.5 Hz) 振荡。这种振荡与心跳形态无关,不会提供有意义的信息 [6]。
设计一个通带频率范围为 [0.5, 40] Hz 的带通滤波器,以消除漂移和任何高频噪声。消除这些分量可改进 LSTM 训练,因为网络不会学习不相关特征。对 tall 数据元胞数组使用 cellfun 来以并行方式对数据集进行滤波。
% Bandpass filter design hFilt = designfilt('bandpassiir', 'StopbandFrequency1',0.4215,'PassbandFrequency1', 0.5, ... 'PassbandFrequency2',40,'StopbandFrequency2',53.345,... 'StopbandAttenuation1',60,'PassbandRipple',0.1,'StopbandAttenuation2',60,... 'SampleRate',250,'DesignMethod','ellip'); % Create tall arrays from the transformed datastores and filter the signals tallTrainSet = tall(trainDs); tallTestSet = tall(testDs); filteredTrainSignals = gather(cellfun(@(x)filter(hFilt,x),tallTrainSet(:,1),'UniformOutput',false));
Evaluating tall expression using the Parallel Pool 'Processes': - Pass 1 of 1: Completed in 19 sec Evaluation completed in 19 sec
trainLabels = gather(tallTrainSet(:,2));
Evaluating tall expression using the Parallel Pool 'Processes': - Pass 1 of 1: Completed in 1.6 sec Evaluation completed in 1.7 sec
filteredTestSignals = gather(cellfun(@(x)filter(hFilt,x),tallTestSet(:,1),'UniformOutput',false));Evaluating tall expression using the Parallel Pool 'Processes': - Pass 1 of 1: Completed in 0.91 sec Evaluation completed in 0.93 sec
testLabels = gather(tallTestSet(:,2));
Evaluating tall expression using the Parallel Pool 'Processes': - Pass 1 of 1: Completed in 0.8 sec Evaluation completed in 0.88 sec
对一种典型情况下的原始信号和滤波后的信号绘图。
trainData = gather(tallTrainSet);
Evaluating tall expression using the Parallel Pool 'Processes': - Pass 1 of 1: Completed in 1.5 sec Evaluation completed in 1.6 sec
figure
subplot(2,1,1)
plot(trainData{95,1}(2001:3000))
title('Raw')
grid
subplot(2,1,2)
plot(filteredTrainSignals{95}(2001:3000))
title('Filtered')
grid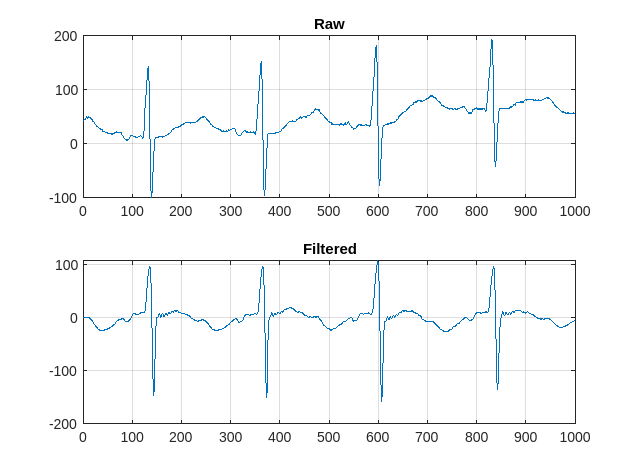
尽管滤波后的信号的基线可能会使习惯于在医疗设备上进行传统 ECG 测量的医生感到困惑,但实际上网络将受益于漂移消除。
使用滤波后的 ECG 信号训练网络
使用与以前相同的网络架构基于滤波后的 ECG 信号训练 LSTM 网络。
if actionFlag == "Train networks" filteredNet = trainnet(filteredTrainSignals,trainLabels,layers,"crossentropy",options); end
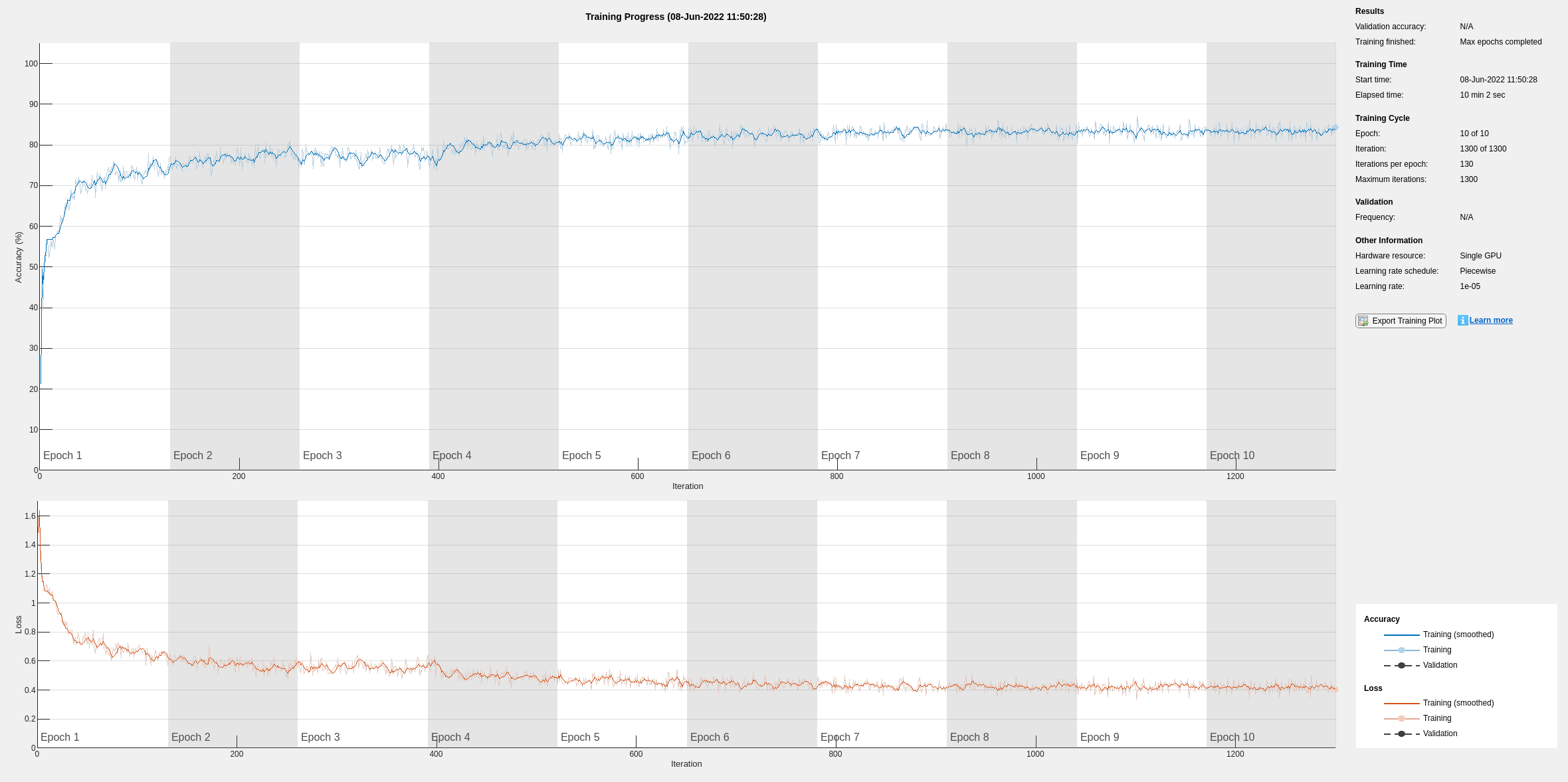
信号预处理将训练准确度提高到 80% 以上。
对滤波后的 ECG 信号进行分类
使用更新后的 LSTM 网络对预处理后的测试数据进行分类。
scores = minibatchpredict(filteredNet,filteredTestSignals,'MiniBatchSize',50,'UniformOutput',false); predFilteredTest = scores2label(scores,classNames);
将分类性能可视化为混淆矩阵。
figure
confusionchart(vertcat(testLabels{:}),vertcat(predFilteredTest{:}),'Normalization','row-normalized');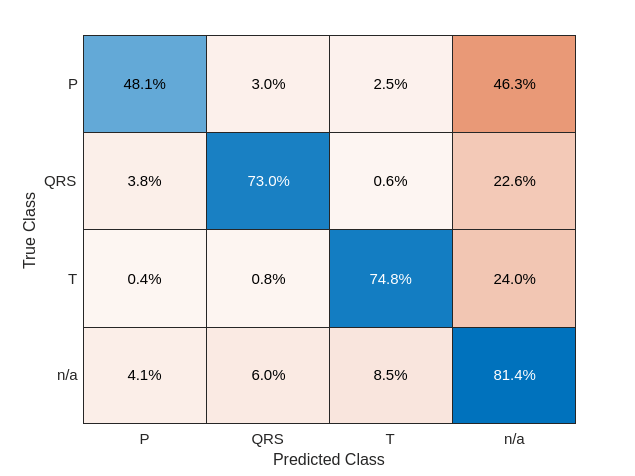
简单的预处理提高了分类性能。
ECG 信号的时频表示
时间序列数据成功分类的常见方法是提取时频特征并将其馈送到网络而不是原始数据。然后,网络同时跨时间和频率学习模式 [7]。
傅里叶同步压缩变换 (FSST) 计算每个信号采样的频谱,因此对于需要保持与原始信号相同的时间分辨率的分割问题,它是可直接使用的理想选择。使用 fsst 函数检查其中一个训练信号的变换。指定长度为 128 的凯塞窗以提供足够的频率分辨率。
data = preview(trainDs);
figure
fsst(data{1,1},250,kaiser(128),'yaxis')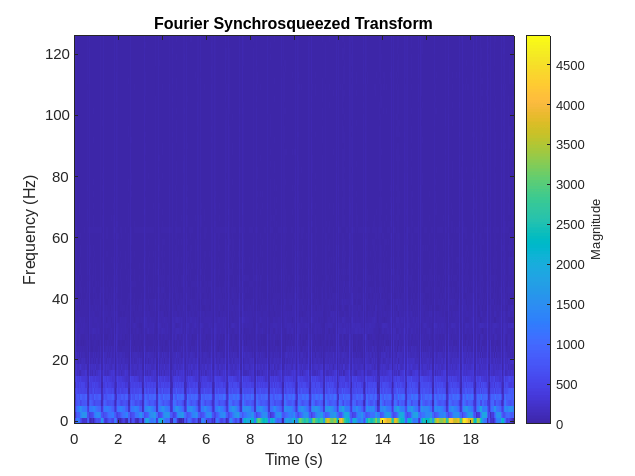
基于感兴趣的频率范围 [0.5, 40] Hz 计算训练数据集中每个信号的 FSST。将 FSST 的实部和虚部视为单独的特征,并将两个分量都馈送到网络中。此外,通过减去均值并除以标准差来标准化训练特征。使用变换后的数据存储、extractFSSTFeatures 辅助函数和 tall 函数来并行处理数据。
fsstTrainDs = transform(trainDs,@(x)extractFSSTFeatures(x,250)); fsstTallTrainSet = tall(fsstTrainDs); fsstTrainData = gather(fsstTallTrainSet);
Evaluating tall expression using the Parallel Pool 'Processes': - Pass 1 of 1: Completed in 47 sec Evaluation completed in 47 sec
对测试数据重复此过程。
fsstTTestDs = transform(testDs,@(x)extractFSSTFeatures(x,250)); fsstTallTestSet = tall(fsstTTestDs); fsstTestData = gather(fsstTallTestSet);
Evaluating tall expression using the Parallel Pool 'Processes': - Pass 1 of 1: Completed in 20 sec Evaluation completed in 20 sec
调整网络架构
修改 LSTM 架构,使网络接受每个采样的频谱,而不是单个值。检查 FSST 的大小以查看频率的数量。
size(fsstTrainData{1,1})ans = 1×2
5000 40
指定一个包含 40 个输入特征的 sequenceInputLayer。保持其余网络参数不变。
layers = [ ... sequenceInputLayer(40) lstmLayer(200,'OutputMode','sequence') fullyConnectedLayer(4) softmaxLayer];
使用 ECG 信号的 FSST 训练网络
使用变换后的数据集训练更新后的 LSTM 网络。
if actionFlag == "Train networks" fsstNet = trainnet(fsstTrainData(:,1),fsstTrainData(:,2),layers,"crossentropy",options); end
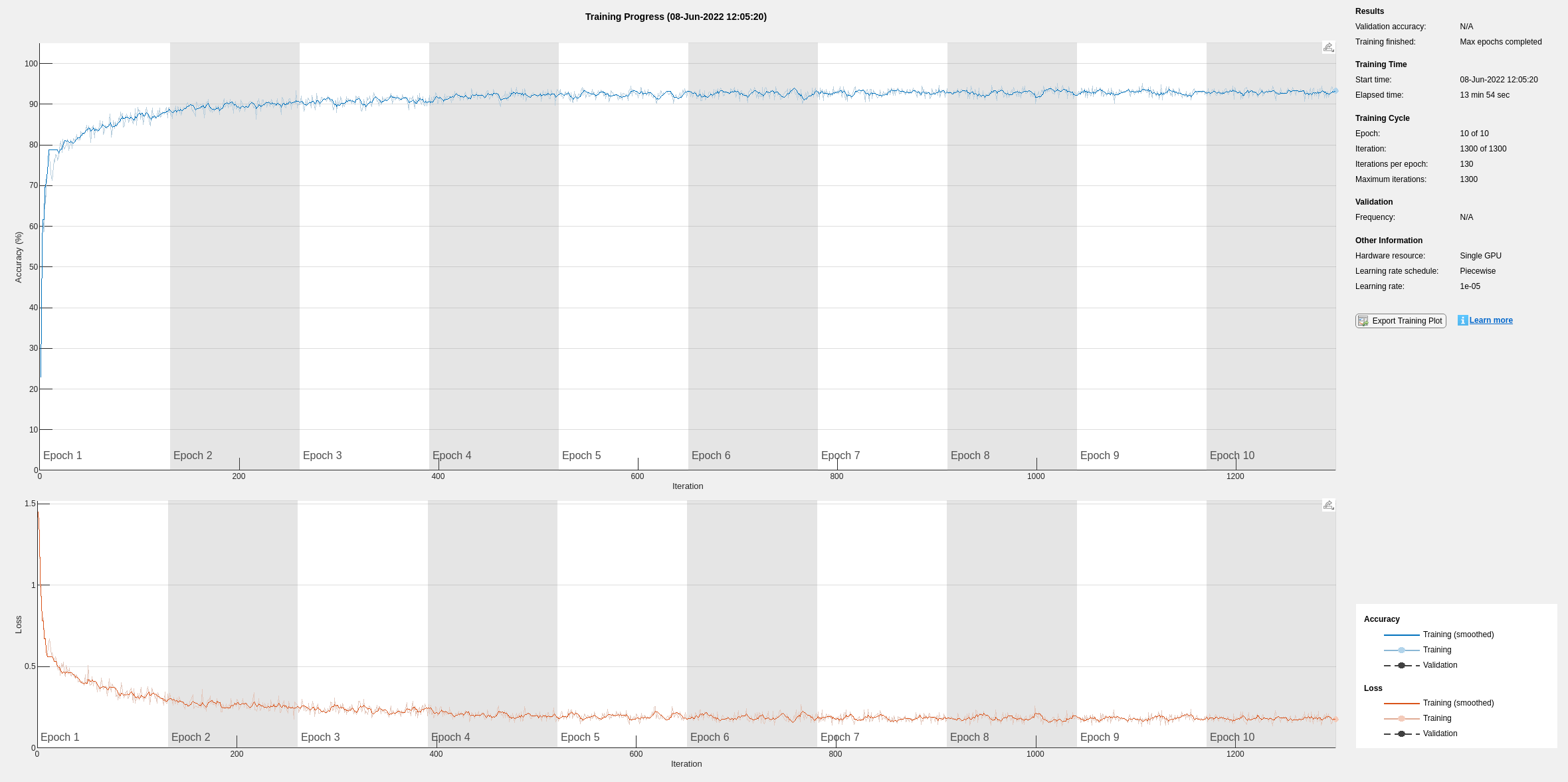
使用时频特征提高了训练准确度,准确度现在已超过 90%。
用 FSST 对测试数据进行分类
使用更新后的 LSTM 网络和提取的 FSST 特征,对测试数据进行分类。
scores = minibatchpredict(fsstNet,fsstTestData(:,1),'MiniBatchSize',50,'UniformOutput',false); predFsstTest = scores2label(scores,classNames);
将分类性能可视化为混淆矩阵。
confusionchart(vertcat(fsstTestData{:,2}),vertcat(predFsstTest{:}),'Normalization','row-normalized');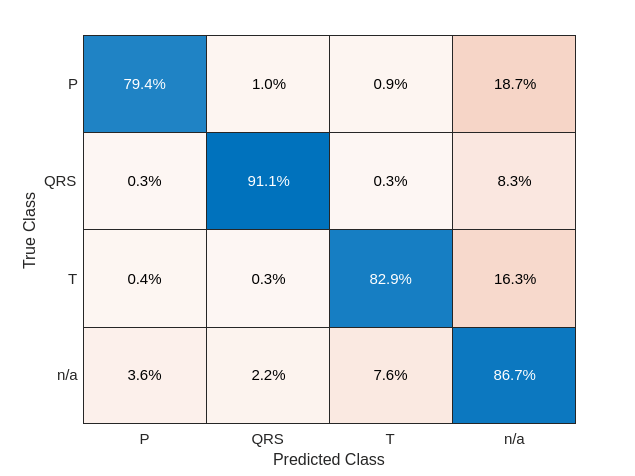
与原始数据结果相比,使用时间频率表示法将 T 波分类提高了约 25%,将 P 波分类提高了约 40%,将 QRS 复合波分类提高了 20%。
使用 signalMask 对象将网络预测值与单个 ECG 信号的真实值标签进行比较。绘制感兴趣的区域时忽略 "n/a" 标签。
testData = gather(tall(testDs));
Evaluating tall expression using the Parallel Pool 'Processes': - Pass 1 of 1: Completed in 0.77 sec Evaluation completed in 0.86 sec
Mtest = signalMask(testData{1,2}(3000:4000));
Mtest.SpecifySelectedCategories = true;
Mtest.SelectedCategories = find(Mtest.Categories ~= "n/a");
figure
subplot(2,1,1)
plotsigroi(Mtest,testData{1,1}(3000:4000))
title('Ground Truth')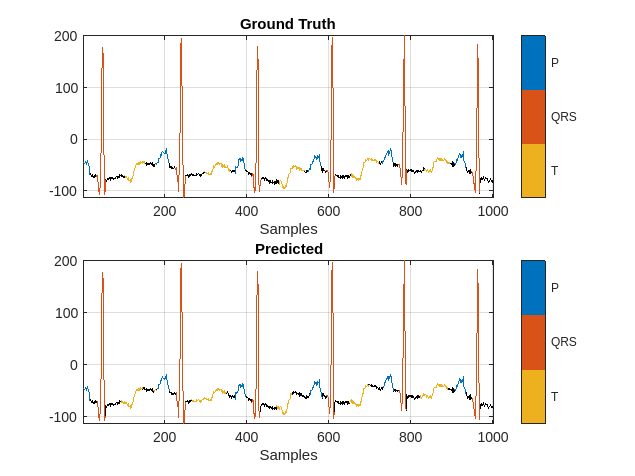
Mpred = signalMask(predFsstTest{1}(3000:4000));
Mpred.SpecifySelectedCategories = true;
Mpred.SelectedCategories = find(Mpred.Categories ~= "n/a");
subplot(2,1,2)
plotsigroi(Mpred,testData{1,1}(3000:4000))
title('Predicted')
结论
此示例说明了信号预处理和时频分析是如何提高 LSTM 波形分割性能的。带通滤波和基于傅里叶的同步压缩使所有输出类的平均改进程度从 55% 提高到了 85% 左右。
参考资料
[1] McSharry, Patrick E., et al."A dynamical model for generating synthetic electrocardiogram signals."IEEE® Transactions on Biomedical Engineering.Vol. 50, No. 3, 2003, pp. 289–294.
[2] Laguna, Pablo, Raimon Jané, and Pere Caminal."Automatic detection of wave boundaries in multilead ECG signals:Validation with the CSE database."Computers and Biomedical Research.Vol. 27, No. 1, 1994, pp. 45–60.
[3] Goldberger, Ary L., Luis A. N. Amaral, Leon Glass, Jeffery M. Hausdorff, Plamen Ch.Ivanov, Roger G. Mark, Joseph E. Mietus, George B. Moody, Chung-Kang Peng, and H. Eugene Stanley."PhysioBank, PhysioToolkit, and PhysioNet:Components of a New Research Resource for Complex Physiologic Signals."Circulation.Vol. 101, No. 23, 2000, pp. e215–e220. [Circulation Electronic Pages; http://circ.ahajournals.org/content/101/23/e215.full].
[4] Laguna, Pablo, Roger G. Mark, Ary L. Goldberger, and George B. Moody."A Database for Evaluation of Algorithms for Measurement of QT and Other Waveform Intervals in the ECG."Computers in Cardiology.Vol.24, 1997, pp. 673–676.
[5] Sörnmo, Leif, and Pablo Laguna."Electrocardiogram (ECG) signal processing."Wiley Encyclopedia of Biomedical Engineering, 2006.
[6] Kohler, B-U., Carsten Hennig, and Reinhold Orglmeister."The principles of software QRS detection."IEEE Engineering in Medicine and Biology Magazine.Vol. 21, No. 1, 2002, pp. 42–57.
[7] Salamon, Justin, and Juan Pablo Bello."Deep convolutional neural networks and data augmentation for environmental sound classification."IEEE Signal Processing Letters.Vol. 24, No. 3, 2017, pp. 279–283.
另请参阅
confusionchart | fsst (Signal Processing Toolbox) | labeledSignalSet (Signal Processing Toolbox) | lstmLayer | trainnet | trainingOptions | dlnetwork